



版权说明:本文档由用户提供并上传,收益归属内容提供方,若内容存在侵权,请进行举报或认领
文档简介
1、全基因组重测序数据分析1. 简介(Introduction)通过高通量测序识别发现 de novo的somatic和germ line突变,结构变异-SNV,包括重 排突变(deletioi n, duplicati on 以及 copy number variatio n )以及 SNP 的座位;针对重排 突变和SNP的功能性进行综合分析;我们将分析基因功能(包括miRNA ),重组率(Recomb in ation )情况,杂合性缺失(LOH )以及进化选择与mutation之间的关系;以及这些关系将怎样使得在disease ( cancer)genome中的mutation产生对应的易感
2、机制和功能。我们将在基因组学以及比较基因组学,群体遗传学综合层面上深入探索疾病基因组和癌症基因组。StmctumVariathnC hroiniicwiial to<n*iobp gen (HU FC>loKPne<irdetectioD.SeqtiEflce VarijifliGni* M KmJMrifelCk IlWtbln 1 Cpjii(CVh J* Ib4vIi* SrfMMLiJ 4v|MMiiiiiii* I rrwai viuIk«« KWi-CW r小命* Mw rad f-kiMihW- « mtmI HfihHAdMMMk
3、rosccppk tad subcbromosoEnjil Q-rn*a*l iaUiiLi BwiB Cl rMWB-tl BrWIlH I- laf rfrsvilv iFim< O.r*BM«ul BbHirKiLhnMolccil&r gtBCilc dt teflon实验设计与样本(1) Case-Control 对照组设计(2 )家庭成员组设计:父母-子女组(4人、3人组或多人); 初级数据分析1.数据量产出:总碱基数量、Total Mapping Reads 、Uniquely Mapping Reads统计,测序深度分析。2 .一致性序列组装:与参考基因
4、组序列(Refere nee gen ome seque nee)的比对分析,禾U用贝叶斯统计模型检测出每个碱基位点的最大可能性基因型,并组装出该个体基因组的一致序列。3. SNP检测及在基因组中的分布:提取全基因组中所有多态性位点,结合质量值、测序深 度、重复性等因素作进一步的过滤筛选,最终得到可信度高的SNP数据集。并根据参考基因组信息对检测到的变异进行注释。4 .In Del检测及在基因组的分布:在进行mappi ng的过程中,进行容 gap的比对并检测可 信的short In Del。在检测过程中,gap的长度为15个碱基。对于每个In Del的检测,至少 需要3个Paired-End
5、 序列的支持。5. Structure Variation 检测及在基因组中的分布:能够检测到的结构变异类型主要有:插入、 缺失、复制、倒位、易位等。根据测序个体序列与参考基因组序列比对分析结果,检测全基 因组水平的结构变异并对检测到的变异进行注释。高级数据分析1.测序短序列匹配(Read Mapping )(1) 屏蔽掉 Y染色体上假体染色体区域(pseudo-autosomal region ),将Read与参考序 列NCBI36进行匹配(包括所有染色体,未定位的con tig,以及线粒体序列 mtDNA (将用校正的剑桥参考序列做替代)。采用标准序列匹配处理对原始序列文件进行基因组匹配,
6、将Read与参考基因组进行初始匹配;给出匹配的平均质量得分分布;(2) 碱基质量得分的校准。我们采用碱基质量校准算法对每个Read中每个碱基的质量进 行评分,并校准一些显著性误差,包括来自测序循环和双核苷酸结构导致的误差。(3) 测序误差率估计。pseudoautosomal con tigs,short repeat regions (包括 segme ntalduplicati on , simple repeat seque nee- 通过 tandem repeat 识另 U算法识另 U) 将被过滤;2. SNP Calling 计算 (SNP Calling )我们可以采用整合多种
7、SNP探测算法的结果,综合地,更准确地识别出SNP。通过对多种算法各自识别的 SNP进行一致性分析,保留具有高度一致性的SNP作为最终SNP结果。这些具有高度一致性的 SNP同时具有非常高的可信度。在分析中使用到的SNP识别算法包括基于贝叶斯和基因型似然值计算的方法,以及使用连锁不平衡 LD或推断技术用于优化SNP识别检出的准确性。统计SNV的等位基因频率在全基因组上的分布稀有等位基因数目在不同类别的 SNV中的比率分布(a) ; SNV的类别主要考虑:(1)无 义(nonsense ) , (2)化学结构中非同义,(3)所有非同义,(4)保守的非同义,(5) 非编码,(6)同义,等类型SNV
8、 ;另外,针对保守性的讨论, 我们将分析非编码区域 SNV 的保守型情况及其分布(图 a, b )ANS o呦輻帅M30252QT5W備 OLoi.olm(>.(>.(>.(>.(>.(>. b ssv Eb- i 33. 短插入 / 缺失探测(Short In sertio n /Deletion(In del ) Call)(1) .计算全基因组的in del变异和基因型检出值的过程计算过程主要包含 3步:(1)潜在的in del的探测;(2)通过局部重匹配计算基因型的似 然值;(3)基于LD连锁不平衡的基因型推断和检出识别。In del在X,Y染色体上
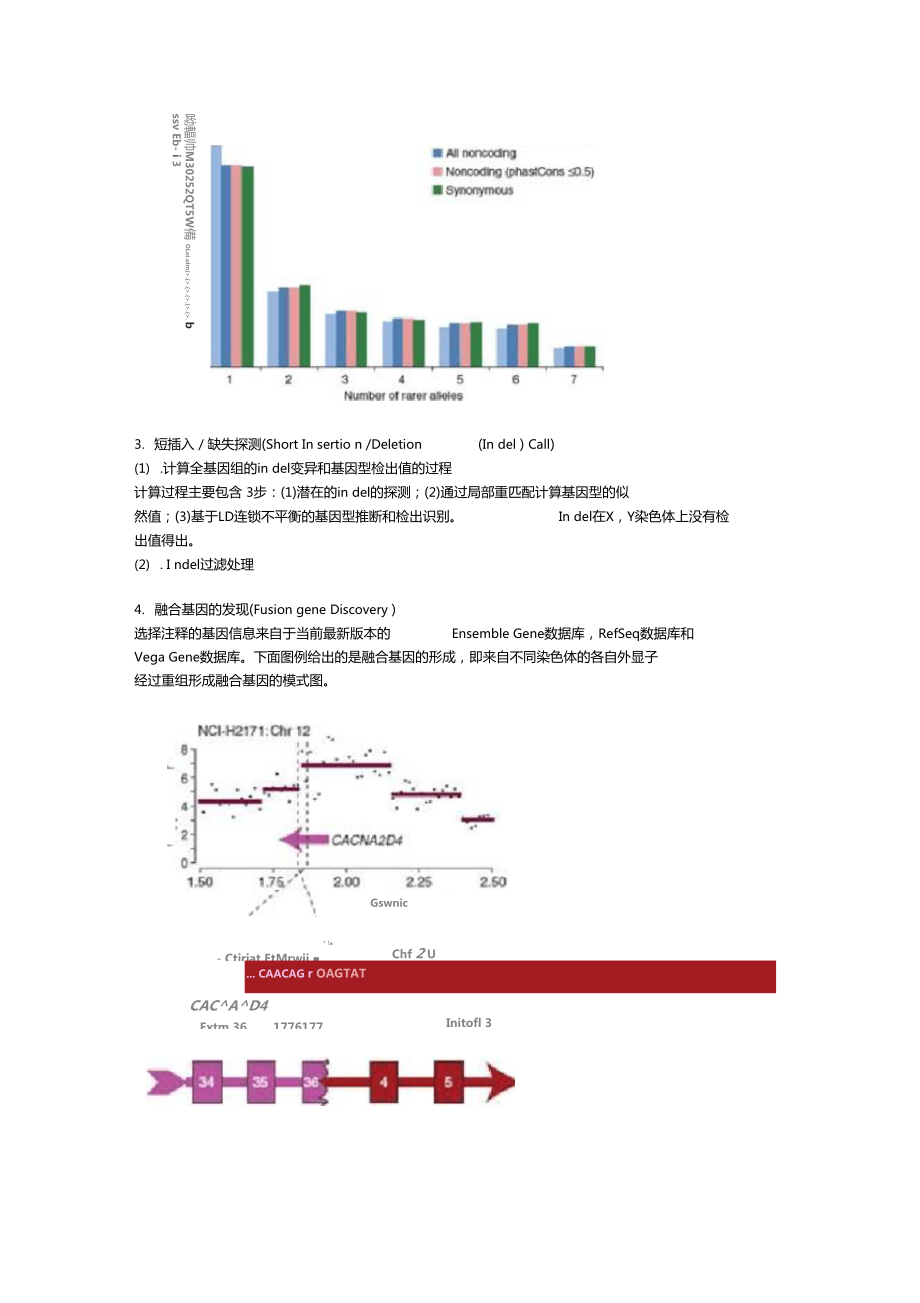
9、没有检出值得出。(2) . I ndel过滤处理4. 融合基因的发现(Fusion gene Discovery )选择注释的基因信息来自于当前最新版本的Ensemble Gene数据库,RefSeq数据库和Vega Gene数据库。下面图例给出的是融合基因的形成,即来自不同染色体的各自外显子 经过重组形成融合基因的模式图。Gswnic 1X4如fl Mb* !- Ctiriat EtMrwjj Chf 2 U. CAACAG r OAGTATCACAD4Extm 361776177Initofl 35. 结构变异(Structure Variation )结构变异(Structure Var
10、iation SV)是基因组变异的一类主要来源,主要由大片段序列(一般 >1kb)的拷贝数变异(copy number variatio n, CNV)以及非平衡倒位(un bala nee in version )事件构成。目前主要一些基因组研究探测识别的SV大约有20,000个(DGV数据库)。在某些区域上,甚至 SV形成的速率要大于 SNP的速率,并与疾病临床表型具有很大关联。我们不仅可以通过测序方式识别公共的SV,也可以识别全新的 SV。全新的SV的生成一般在germ line和突变机制方面都具有所报道。然而,当前对SV的精确解析需要更好的算法实现。同时,我们也需要对SV的形成机
11、制要有更重要的认知,尤其是 SV否起始于祖先基因组座位的插入或缺失,而不简单的根据等位基因频率或则与参考基因组序列比对判断。SV的功能性也结合群体遗传学和进化生物学结合起来,我们综合的考察SV的形成机制类别。SV形成机制分析,包括以下几种可能存在的主要机制的识别发现:(A)同源性介导的直系同源序列区段重组(NAHR);(B )与DNA双链断裂修复或复制叉停顿修复相关的非同源重组(NHR);(C) 通过扩展和压缩机制形成可变数量的串联重复序列(VNTR);(D) 转座元件插入(一般主要是长/短间隔序列元件LINE/SINE或者伴随TEI相关事件 的两者的组合)。结构变异探测和扩增子(Amplic
12、on )的探测与识别分析:如下图所示6. 测序深度分析测序深度分析就是指根据基因组框内覆盖度深度与期望覆盖度深度进行关联,并识别出SV。我们也将采用不同算法识别原始测序数据中的缺失片段(deletion )和重复片段(duplication )。7. SV探测识别结果的整合与FDR推断(可选步骤)(1). PCR或者芯片方式验证 SV(2).计算FDR-错误发现率(配合验证试验由客户指定)筛选SV检出结果用于SV的合并和后续分析:我们通过不同方式探测识别SV的目的极大程度的检出SV,并且降低其 FDR( <=10% )。通过下属筛选方法决定后续分析所使用到的SV集合。每种SV探测识别算法
13、得到的SV的FDR要求小于10%,并将各自符合条件的SV合并;对于FDR大于10%的算法计算识别的 SV结果,如果有PCR和芯片平 台验证数据,同样可以纳入后续SV分析中。最后,针对不同算法得到的SV,整合处理根据breakpoint断点左右重合覆盖度的置信区间来评定;8. 变异属性分析(1) n eutral coalesce nt 分析测序数据可以探测到低频率的变异体(MAF<=5% )。根据来自群体遗传学理论(neutralcoalesce nt理论)的期望值可以计算低频度变异的分布。我们用不同等位基因频率下每Mb变异数目与n eutral coalesce nt选择下的期望值比值
14、,即每Mb基因组win dows内的theta观测值,来刻画和反映自然纯化选择与种群(cancer cell-line可以特定的认为是可以区分的种群)增长速率。该分布分别考察SNP (蓝色线),In del (红色线),具有基因型的大片段缺失(黑色线),以及外显子区域上的SNP (绿色线)在不同等位基因频率区间上的theta情况(参见下图)。Variant allele frequency(2).全新变异体(novel variant)的等位基因频率和数量分布分析对象包括全新预测的SNP , in del , large deletio n,以及外显子 SNP在每个等位基因频率类别下的数目比率
15、(fraction )(参见下图);全新预测是指预测分析结果与dbSNP (当前版本129 )以及deletion数据库dbVar ( 2010年6月份版本)和已经发表的有关 in dels 研究的基因组数据经过比较后识别确定的全新的SNP,in del以及deletion。dbSNP包含SNP 和 in dels; dbVAR 包含有 deletio n, duplicatio n, 以及 mobile eleme nt in sertio n。dbRIP 以及其他基因组学研究(JC Ventrer以及Watson基因组,炎黄计划亚洲人基因组)结果 提 供 的shortin dels禾口la
16、rgedeleti on。-®>oc upoelr6 4 2 o,a a0.0-0.0 0.2 04 0.6 0.8 1-0Variant allele frequency(3) .变异体的大小分布以及新颖性分布计算 SNP , Deletion,以及 Insertion 大小分布;计算 SNP , Deletion,以及 Insertion 中属 于全新预测结果的数目占已有各自参考数据库数目的比例(相对于dbSNP数据库;dbSNP包含 SNP 和 indels;dbVAR 包含有 deletion,duplication, 以及 mobile element inserti
17、on。 dbRIP以及其他基因组学研究(JC Ventrer以及 Watson基因组,炎黄计划亚洲人基因组)结果提供的short in dels和large deletion )其中,可以给出 LINE,Alu的特征位置。37G543?1O 匸也AQgain仝孑100kbkb “OtvipObp 10 kh3 2 10 o o o O6-ODeletionsSNPs Insartions<-og1D阿(4) .结构变异SV的断点联结点(BreakPoint Junction) 分析根据SV不同检出结果经过一些列筛选步骤构建所有结构变异SV的断点联结点数据库,保留长度大于等于50bp的SV
18、 ;分析断点联结点处具有 homology或者microhomology的SV ; 并将同一染色体,起始和终止位置坐标下的不同SV进行去冗余处理。分析识别SV的断点联结点(Breakpoint):将Breakpoint按照可能形成的方式可以分类为 以下几类:(a) 非等位基因同源重组型(non-allelic homologous recombination-NAHR);(b) 非同源重组 (non homologous recomb in atio n-NHR),包括 non homologous en d-jo ining (NHEJ)和 fork stalling /template s
19、witching ( FoSTeS/MMBIR );(c)可变串联重复(VNTR)(d)转座插入元件(TEI )。313V512IInsertion traceFomwaiofi mechanism stacked hts-tognvnboogrannBMAHR DBNHR (3 TEIVWTR10飞图CSV形成偏好性分析分析SV形成机制与断裂点临近区域序列的关系,包括染色质界标(端粒,中心粒),重组 高发热点区域,重复序列以及GC含量,短DNA motif和微同源区域(microhomologyregion )。D«tan» tQ lelomeFiM1,206a.e Oe
20、+O7 -C.Oa-i-OC-1 0fl+D8 rDtstsnce to eerrtncfnereeNAUR NHR TEIDe-tanw tn 穷用eny txwdwn1,2»+W-w-U NAH Fl NHR TEId.4.3 o ovlluQAA15co+ NHRBackground -p- Expectation0510152025Lenqfri ol microhomoloq (bp)9. 突变率估计针对以家庭成员为单位的测序方案,我们主要探测de novo的突变(DNM );通过采用不同的方法/算法,我们给出每个家庭一份推断的DNM报表;(1) 根据基因型推断结果,分别对
21、每人每碱基位置上的de novo突变进行综合度量;(2) 采用贝叶斯方法计算家庭组设计中DNM的后验概率10. SNP,SNV功能分析与注释(1).祖先等位基因的注释通过将人类(NCBI36),黑猩猩(chimpanzee2.1 ),猩猩(PPYG2)以及恒河猴(MMUL1) 4种基因组进行基因组比对,发现保守的序列区域,计算祖先等位基因;以及 duplicatio n/deleti on 事件的进化分析。(2).分析基因结构序列上不同区域的多样性( Diversity )与分歧进化(diverge nee )根据基因型分析结果计算基因结构序列上的多样性程度,即杂合度(heterozygosi
22、ty);杂合度指标可以说明选择效应的存在以及局部变异的结构分布特征模式。我们将考虑基因5' UTR上游200bp,5' UTR,第一个外显子,第一个内含子,中间外显子,中间内含子,最末外显子和内含子,以及 3' UTR及其下游200bp区域左右考察的范围(参见下图a)。分析编码 转录本的起始/终止位置临近区域的多样性和进化分歧度(参见下图 b)。0.UU120.00060.0000Jd0.016-0.012'0 008-0.4 -0.2 0.0 0.2 04cM from transcription start/stop(3).疾病变异体探测将样本测序中分析得到
23、 SV与HGMD疾病变异体数据进行比对,得到交叉记录的错义和无 义的SNP ;通过将HGMD疾病关联突变与 CUI (疾病概念分类标识数据库) 比对获得HGMD 中所有SV的疾病表型,并获得HGMD与测序数据分析得到的 SV的疾病表型;并通过Fisher 检验和Bonferroni多重假设检验校正计算样本SV所富集的疾病表型。pHddw(4).拷贝数变异CNV所含基因的功能注释将CNV是否覆盖区段重复 SD区域分类为2大类, 计算,显著性在横轴表示;各种显著性功能在纵轴表示。每类CNV的所含基因的功能富集情况CAH/l iwi tswrbppmg 5Ca如屮冲吕站-LogJPrthjPi0E初
24、PmiMHMftBM(5) .变异的功能性分析与注释(a) . SNP, Indels以及大的结构变异 SV的功能注释;(b) .对包含翻译起始注释信息的转录本编码区上的SNP分类为:同义SNP ,非同义SNP 和无义SNP (引入终止子),干扰终止子的 SNP,以及干扰剪接位点的 SNP ;为了降低假 阳性,我们采用严格的筛选方式过滤来自 in dels的错误;(c) 对错义编码区突变的功能性分析:通过信息学分析算法评估相对于生殖系变异的体细 胞突变对蛋白质的结构和功能的影响效应。(6). SNV,SNP与miRNA研究之间的关联分析miRNA是起重要的调控作用的小分子,我们将对miRNA的
25、pri-mRNA ,pre-miRNA 以及miRNA靶基因序列进行分析,识别潜在的SNP功能位点。据文献研究提供证据表明Humanpre-miRNA的二级结构中存在不同位置上的SNP,我们将通过热力学稳定性分析方法评估SNP对pre-miRNA结构的影响;另外,我们也将对miRNA-Target靶基因相互作用位点做分析,评估对SNP对靶基因靶向性的影响。MIR*19MIR(7). SNV,SNP与GWAS研究之间的关联分析分析GWAS研究中得到的易感基因在基因组上不同坐标上的OR值分布情况;将当前已知的GWAS研究成果与SNP进行比较;根据 LD连锁不平衡将SNP与易感基因的关系进 行深入讨
26、论;直接与间接关联方法可以分别识别与表型相关的SNP,对于不易获得(missing)和定位的SNP,通过LD连锁不平衡推断疾病易感基因突变座位。(8)生物学通路(代谢通路,信号通路)分析生物学通路(Biological pathway ),包括代谢通路和信号转导通路是生物功能的重要组成 部分,我们将各种形式的突变、变异,包括SNV和SNP,的对应基因放到生物学通路中进行综合分析,考察功能性突变对pathway的影响程度和影响的规律。通过GSEA (配合芯片表达谱数据),KS检验,超几何分布检验等方法对变异基因在某些pathway的富集程度进行排序,识别发生功能改变的潜在通路。(9).蛋白质-蛋
27、白质相互作用(PPI )网络分析蛋白质相互作用也是生物分子功能增益和缺失的重要途径,因此我们针对蛋白质相互作用网络中的突变的蛋白及其收到影响的网络节点蛋白进行系统分析,并对收到影响的网络子结构进行功能注释分析和聚类富分析。我们采用网络分析算法对由于各种突变所受到影响的子网 络(sub network )进行功能富集度的分析;3(10).顺式基因调控网络模块(CRM )分析(a) 启动子序列分析包括动子区域上的 Motif预测,并与已知转录因子数据库 TRANSFAC和JASPAR中的TFBS 结合位点进行比对;启动子区域上保守性分析,分析突变位置和保守性区域的关联;(b) 计算全基因组保守性。
28、确定TFBS的保守性以及 mutation位置的保守性;(11 )重排(arrangements )与突变(mutation )的全基因组统计(a) .体细胞(somatic)和生殖系(germline )重排(arrangements )体细胞突变是相对于germ line突变的一类需要重要分析的内容,我们针对Case-control设计的测序方案可以分别分析突变的情况,包括SNV , in del,以及 CNV ;如果仅在tumor/disease(Case 组)出现而不在normal (对照组)出现的突变我们可以认为是somatic体细胞突变。将somatic mutation 与dbS
29、NP数据库比对可以发现潜在的全新的突变和有记 录的突变位置。然后,将突变分别比对到基因区域和非基因区域。基因区域具体包括:内含子区,UTR,剪接位点区和外显子区。其中外显子区分别统计:同义(synonymous ),缺失(deletion ),阅读框移位 (frameshift ),插入(insertion ),错义(missense ),无义(nonsense ) 以及非编码蛋白外显子 (non-protein coding exon)等不同类型。综合不同方面分析的结果,并按照突变分类给出各重排(arrangements)类型:SNV , CNV的数目统计数据表(参见下图)。对每一测序样本分别进行标注,包括体细胞突
温馨提示
- 1. 本站所有资源如无特殊说明,都需要本地电脑安装OFFICE2007和PDF阅读器。图纸软件为CAD,CAXA,PROE,UG,SolidWorks等.压缩文件请下载最新的WinRAR软件解压。
- 2. 本站的文档不包含任何第三方提供的附件图纸等,如果需要附件,请联系上传者。文件的所有权益归上传用户所有。
- 3. 本站RAR压缩包中若带图纸,网页内容里面会有图纸预览,若没有图纸预览就没有图纸。
- 4. 未经权益所有人同意不得将文件中的内容挪作商业或盈利用途。
- 5. 人人文库网仅提供信息存储空间,仅对用户上传内容的表现方式做保护处理,对用户上传分享的文档内容本身不做任何修改或编辑,并不能对任何下载内容负责。
- 6. 下载文件中如有侵权或不适当内容,请与我们联系,我们立即纠正。
- 7. 本站不保证下载资源的准确性、安全性和完整性, 同时也不承担用户因使用这些下载资源对自己和他人造成任何形式的伤害或损失。
最新文档
- 《富饶的西沙群岛》教学课件-3
- 河北省唐山市遵化一中2025届高三第一次调研考试数学试题文试题
- 《他得的红圈圈多》课件-1
- 财务管理体系培训
- 湖南省郴州市桂阳三中、资兴招飞安仁招飞三校联考2024-2025学年七年级下学期4月月考语文试题(原卷版+解析版)
- DB11 T 384.7-2009 图像信息管理系统技术规范 第7部分 工程要求与验收
- 林业科技培训课件
- 药学库房管理课件
- 二零二五版大学生实习协议书模板
- 车间管理制度几个不准
- 吉林省吉林市2024-2025学年高三下学期3月三模试题 政治 含答案
- 湖北省圆创教育教研中心2025届高三三月联合测评物理试题及答案
- 五下语文期中复习知识点
- 城市轨道交通轨道设备运营维保方案终稿
- 陈仓《我有一棵树》阅读答案
- 铜绞线接地施工方案
- 山东省青岛市市南区2024-2025学年上学期九年级期末质量监测九年级数学试题 (原卷版+解析版)
- 2025年开封大学单招职业适应性测试题库新版
- 中央2025年中国日报社及所属事业单位招聘国内高校应届生笔试历年参考题库附带答案详解
- GB/T 20972.2-2025石油天然气工业油气开采中用于含硫化氢环境的材料第2部分:抗开裂碳钢、低合金钢和铸铁
- 《云南烟草行业》课件

评论
0/150
提交评论